Multiplex real-time PCR phát hiện đồng thời SARS-COV-2 và các biến thể lây truyền nhanh hiện đang lưu hành
Mục tiêu nghiên cứu: Thiết kế một bộ xét nghiệm sử dụng phương pháp multiplex real-time PCR thực hiện được tại các phòng thí nghiệm chẩn đoán để phát hiện 4 biến thể Alpha, Beta, Gamma và Delta cùng hai đột biến giúp virus lây lan nhanh (D614G) và có thể trốn thoát hoạt động của kháng thể đặc hiệu (E484K).
Van H. Pham*1, Thanh V. Nguyen2, Ngọc V. Tran2, Sy N. Dinh3, Nhung V. Nguyen2, Chien D. Vo4, Ha M. Nguyen4,5, Binh T. Pham6, Sa H. Le1, Duy K. Trần1, Quoc V. Nguyen1, Viet Q. Tran1, Huong T. Pham5, Son T. Pham7,
Nam H. D. Nguyen8
1Nam Khoa Biotek,2Hoi Phoi Vietnam, 3Hoi Y Hoc Vietnam, 4Nguyen Tri Phuong Hospital,
5Pham Ngọc Thach Medical University, 6University of Medicine and Pharmacy in HCMC, 7Sydney University,
8Phan Chau Trinh University
Tóm tắt:
Đặt vấn đề: Hiện nay trên thế giới có nhiều biến thể của SARS-CoV-2, trong số đó có 4 biến thể là Alpha (B.1.1.7), Beta (B.1.351), biến thể Gamma (P.1), và biến thể Delta (B.1.617.2) có tốc độ lây lan nhanh hơn chủng ban đầu theo thứ tự là 82%, 161%, 50% và198%. Để phát hiện được các biến thể SARS-CoV-2 đang lưu hành tại một vùng dịch tễ nào đó thì phương pháp mà các nhà nghiên cứu hiện đang sử dụng là giải mã toàn bộ gen của các virus phát hiện được trong các mẫu thử. Phương pháp này có một lợi thế là phát hiện được toàn bộ các đột biến trong bô gen của virus. Ngoài ra phương pháp này còn có thể phát hiện được các đột biến mới và như vậy là có thể phát hiện được các biến thể mới của virus. Tuy nhiên phương pháp giải trình tự có hạn chế là không thể áp dụng được tại các phòng xét nghiệm chống dịch mà chỉ có thể áp dụng được tại các trung tâm có phương tiện giải trình tự mà thôi.
Mục tiêu nghiên cứu: Thiết kế một bộ xét nghiệm sử dụng phương pháp multiplex real-time PCR thực hiện được tại các phòng thí nghiệm chẩn đoán để phát hiện 4 biến thể Alpha, Beta, Gamma và Delta cùng hai đột biến giúp virus lây lan nhanh (D614G) và có thể trốn thoát hoạt động của kháng thể đặc hiệu (E484K).
Phương pháp nghiên cứu: Các primer và probe để phát hiện các biến thể Alpha, Beta, Gamma và Delta được thiết kế dựa trên sự phát hiện các đột biến chuyên biệt của các biến thể này. Các biến thể Alpha, Beta và Delta được phát hiện dựa ARMS Taqman real-time PCR (ARMS: Amplification Refractory Mutation System) với nguyên tắc là nếu có đột biến thì Taqman probe sẽ không bị thủy giải và sẽ không có tín hiệu khuếch đại và ngược lại nếu không có đột biến thì Taqman probe sẽ bị thủy giải và sẽ có tín hiệu khuếch đại. Biến thể Gamma và các đột biến D614G cũng như đột biến E484K được phát hiện dựa trên SNP Taqman real-time PCR với nguyên tắc là mỗi đột biến sẽ được phát hiện bằng hai Taqman probe có reporter khác nhau là FAM và HEX hay TexasRED và CY5 và tùy thuộc vào sự phát huỳnh quang sớm hay trễ của hai Taqman probe này mà kết luận được là có hay không có đột biến. Bộ xét nghiệm được thiết kế gồm ba RT multiplex real-time PCR với multiplex A (MPL-A) phát hiện SARS-CoV-2 dựa trên gene E với mồi và Taqman probe (FAM) theo thiết kế của WHO, biến thể Alpha (HEX) và chứng nội là gene RNAseP (CY5); MPL-B phát hiện biến thể Delta (FAM), biến thể Beta (HEX), và biến thể Gamma (TexasRED/CY5); MPL-C phát hiện D614G (FAM/HEX) và E484K (TexasRED/CY5). Các multiplex được được pha từ AgPath-ID™ One-Step RT-PCR (ThermoFisher, Mỹ). Để kiểm tra các thiết kế mồi và probe dành cho các đột biến có hoạt động hiệu quả không và độ nhạy in-vitro, các trình tự DNA tương ứng cho các đột biến cũng được thiết kế để làm vai trò chứng [+]. Bộ xét nghiệm sau đó được thử trên các mẫu thật là các tách chiết RNA đã có kết quả [+] SARS-CoV-2 được cung cấp từ phòng thí nghiệm có chức năng xét nghiệm.
Kết quả: Thử nghiệm trên các chứng [+] cho kết quả giới hạn phát hiện của gene E và biến thể Alpha là 10-6 fm/µl, biến thể Delta là 10-5 fm/µl, biến thể Beta và biến thể Gamma là 10-7 fm/µl , các đột biến D614G và E484G là 10-5. Không có hiện tượng phát hiện chéo các đột biến hay các biến thể. Thử nghiệm trên các tách chiết RNA đã xác định [+] SARS-CoV-2. Với các mẫu lấy từ TP. HCM, kết quả cho thấy tháng 4/2020 (1 mẫu) là chủng nguyên thủy chưa có đột biến D614G và E484K, trong khi tháng 6 năm 2021 thì các mẫu thử (12) là biến thể Delta có thêm đột biến D614G nhưng không có đột biến E484K. Với các mẫu lấy từ Quảng Nam, kết quả cho thấy vào tháng 6/2020 vừa có chủng có đột biến D614G (3) vừa có chủng nguyên thủy (2), trong khi tháng 6/2021 thì tất cả các chủng là biến thể Alpha (4) và có đột biến D614G. Mẫu có chủng nguyên thủy, mẫu có biến thể Delta, mẫu có biến thể Alpha và mẫu chỉ có đột biến D614G đều được giải trình tự gen S và kết quả hoàn toàn phù hợp với kết quả real-time PCR.
Kết luận: Theo qui luật tiến hóa thì một biến thể có tốc độ lây lan nhanh sẽ dần dần thay thế chủng hoang dại ban đầu và một khi địa phương đạt miễn dịch cộng đồng đối với một biến thể này thì lại có thể nhạy cảm với một biến thể khác do vậy mà có thể bị thay thế bởi một biến thể khác. Chính vì vậy việc triển khai xét nghiệm multiplex real-time PCR để phát hiện 4 biến thể lây lan nhanh tại các phòng xét nghiệm chẩn đoán là rất cần thiết. Với một số kết quả thử trên các mẫu tách chiết RNA, có thể kết luận rằng tại TP. Hồ Chí Minh lúc mới bắt đầu dịch thì SARS-CoV-2 vẫn là chủng hoang dại ban đầu, nhưng hiện nay thì đã hoàn toàn bị thay thế bởi biến thể Delta. Tại Quảng Nam thì khởi đầu dịch là chủng hoang dại nhưng cũng đã lưu hành chủng có đột biến D614G, hiện nay thì biến thể Alpha đang lưu hành và có đột biến D614G. Riêng đột biến E484K thì cho đến hiện nay vẫn chưa xuất hiện và đây là một dấu chỉ cho thấy SARS-CoV-2 chưa kháng được các kháng thể đặc hiệu vùng nhận diện thụ thể trên protein gai của virus.
Từ khóa: SARS-CoV-2 variants, RT Multiplex real-time PCR
Đặt vấn đề
Trong quá trình lây truyền trên người, SARS-CoV-2 đã hình thành nên các đột biến để virus ngày càng lây truyền dễ dàng hơn và theo qui luật tiến hóa thì các virus lây truyền càng nhanh càng dễ thì sẽ dần dần thay thế các virus lây truyền chậm hơn trước đó. SARS-CoV-2 gây bệnh đầu tiên tại Vũ Hán là dòng nguyên thủy tuy nhiên sau khi lan qua Âu Châu thì virus đã có biến đổi đầu tiên trên gen S hình thành nên đột biến D614G, tại vị trí codon 614 của gen S thay vì là aspartic acid (D) lại bị thay thế bằng Glycine (G) làm cho vùng bám thụ thể (RBD) của protein gai mở rộng ra hơn nên giúp cho virus bám vào thụ thể ACE2 dễ hơn và chặt hơn[1]. Chính vì vậy mà biến chủng này, gọi là chủng G, đã thay thế hoàn toàn chủng D trước đó. Thật ra thì đột biến D614G đã xuất hiện rải rác tại Vũ Hán nhưng khi lan qua Châu Âu thì tỷ lệ xuất hiện đã tăng lên đến 20% các chủng và đến tháng 6 đã chiếm 100% các chủng với hậu quả là tạo ra các làn sóng lây nhiễm không chỉ tại Mỹ, các nước Châu Âu mà rất nhiều nước trên thế giới[1]. Tại Việt Nam, SARS-CoV-2 mang đột biến D614G đã xuất hiện rất sớm từ tháng 3 năm 2020 và nguồn gốc là từ các khác du lịch từ Âu Châu[2,3] và vụ dịch xãy ra tại Đà Nẵng vào tháng 7 năm 2020 cũng là do SARS-CoV2 mang đột biến này gây ra.
Nhờ giải trình tự bộ gen của SARS-CoV-2 liên tục mà cho đến hiện nay các nhà khoa học đã phát hiện ra nhiều biến thể của virus đang lưu hành với xuất phát ban đầu có thể từ một số quốc gia[3]. Đó là biến thể Alpha (còn gọi là B.1.1.7) xuất phát từ Anh Quốc được báo cáo từ tháng 12 năm 2020[4], lây lan nhanh hơn 82% so với virus ban đầu; Biến thể Beta (B.1.351) xuất phát từ Nam Phi báo cáo đầu tiên từ tháng 1/2021 có tốc độ lây lan nhanh hơn 50%[5]; Biến thể Gamma (P.1) xuất phát từ Brazil từ tháng 1/2021[6,7] có tốc độ lây lan nhanh hơn 161%; Biến thể Epsilon (B.1.429 và B.1.427) xuất phát từ Hoa Kỳ từ tháng 3/2020[8] có tốc độ lây lan nhanh hơn 20%; Biến thể Delta (B.1.617.2) xuất phát từ Ấn Độ và được báo cáo vào tháng 5 năm 2021[9] có tốc độ lây lan nhanh nhất, hơn 198% so với chủng ban đầu và biến thể này chính là biến thể không chỉ hoành hành tại Ấn Độ mà cả các quốc gia đông Nam Á hiện nay. Ngoài ra còn có các biến thể khác nữa như Kappa (B.1.617.1) xuất phát từ Ấn độ và Eta (B.1.525) xuất phát từ Nigeria hiện đang được điều tra thêm. Các biến thể này có một đặc điểm chung là đều mang đột biến D614G[3] làm cho vùng bám thụ thể trên protein gai của virus mở rộng ra hơn, nhờ vậy mà virus dễ dàng lây nhiễm hơn. Ngoài đột biến D614G, các biến thể còn có thể thêm một số đột biến trên một số codon ảnh hưởng trực tiếp hay gián tiếp lên lên cấu trúc vùng bám thụ thể của protein gai của virus và chính các đột biến này đã làm tăng khả năng lây nhiễm (như đột biến N501Y, E484K…) hay đột biến có thể làm làm gia tăng nguy cơ virus trốn thoát miễn dịch bảo vệ của người do chủng ngừa hoặc kháng lại hiệu quả huyết thanh điều trị[3] (đột biến E484K, K417N/T, L452R, …).
Để phát hiện được các biến thể SARS-CoV-2 đang lưu hành tại một vùng dịch tễ nào đó thì phương pháp mà các nhà nghiên cứu hiện đang sử dụng là giải mã toàn bộ gen của các virus phát hiện được trong các mẫu thử[10]. Phương pháp này có một lợi thế là phát hiện được toàn bộ các đột biến trong bộ gen của virus, không chỉ ở gen S mà còn trên các gen khác như gen N. Không chỉ vậy phương pháp này còn có thể phát hiện được các đột biến mới và như vậy là có thể phát hiện được các biến thể mới của virus. Tuy nhiên phương pháp giải trình tự có hạn chế là không thể áp dụng được tại các phòng xét nghiệm chống dịch mà chỉ có thể áp dụng được tại các trung tâm có phương tiện giải trình tự mà thôi. Để có thể đáp ứng được kịp thời khả năng chống dịch thì phải có giải pháp phát hiện và định lượng SARS-CoV-2 đồng thời phát hiện các biến thể có nguy cơ lây lan cao hiện diện trong mẫu thử lấy từ các trường hợp nhiễm bệnh cũng như có nguy cơ nhiễm bệnh và giải pháp này phải có thể thực hiện được tại các phòng thí nghiệm chống dịch hiện nay, tức là các phòng thí nghiệm có trang bị phương tiện real-time PCR[10].
Mục tiêu nghiên cứu
Mục tiêu chính của đề tài này là nghiên cứu chế tạo bộ xét nghiệm real-time PCR vừa phát hiện và định lượng SARS-CoV-2; Đồng thời phát hiện các biến thể có khả năng lây lan nhanh bao gồm biến thể Anh Quốc (Alpha), biến thể Nam Phi (Beta), biến thể Brazil (Gamma) và biến thể Ấn Độ (Delta); Ngoài ra phát hiện thêm hai đột biến quan trọng liên quan nhiều đến khả năng lây lan (D614G) và nguy cơ giảm hiệu quả miễn dịch bảo vệ của vaccin (đột biến E484K).
Mục tiêu cụ thể của đề tài là: (1) Xác định độ nhạy in-vitro của bộ xét nghiệm tức là giới hạn phát hiện chính xác các mục tiêu đích trên các mẫu chứng dương được thiết kế. (2) Xác định độ nhạy và độ đặc hiệu khi thử nghiệm trên các mẫu thử thật là các tách chiết RNA cũng như một số mẫu thật đã xác định [+] với SARS-CoV-2 đồng thời so sánh với kết quả giải trình tự để xác định độ đặc hiệu của bộ xét nghiệm.
Phương pháp nghiên cứu
1. Thiết kế và chế tạo bộ xét nghiệm real-time PCR
Sử dụng các mồi và probe phát hiện gen E của virus do WHO công bố để phát hiện và định lượng SARS-CoV-2,. Để phát hiện các biến thể, chúng tôi lựa chon các đột biến đặc hiệu[3] cho biến thể Alpha, Beta, Gamma và Delta rồi thiết kế các mồi và probe đặc hiệu các đột biến này. Cũng thiết kế thêm các mồi và probe đặc hiệu để phát hiện hai đột biến D614G và E484K. Các biến thể Alpha, Beta và Delta được phát hiện dựa vào ARMS Taqman real-time PCR (ARMS: Amplification Refractory Mutation System) haychính xac hơn Taqman real-time PCR thất bại đích[10] (Target Failure Taqman Real-time PCR) với nguyên tắc là nếu có đột biến thì Taqman probe sẽ không bị thủy giải và sẽ không có tín hiệu khuếch đại và ngược lại nếu không có đột biến thì Taqman probe sẽ bị thủy giải và sẽ có tín hiệu khuếch đại. Biến thể Gamma và các đột biến D614G cũng như đột biến E484K được phát hiện dựa trên SNP Taqman real-time PCR[10] với nguyên tắc là mỗi đột biến sẽ được phát hiện bằng hai Taqman probe có reporter khác nhau là FAM và HEX hay TexasRED và CY5 và tùy thuộc vào sự phát huỳnh quang sớm hay trễ của hai Taqman probe này mà kết luận được là có hay không có đột biến. Ngoài ra mồi và probe đặc hiệu gene RNAseP của tế bào người cũng được sử dụng để làm chứng nội. Các chứng dương (C[+]) là các trình tự có đột biến và trình tự không đột biến tương ứng, gồm các loại sau đây: 1 chứng dương cho gen E (E_Oligo); 2 chứng dương cho biến thể Alpha (UK_Oligo và notUK_Oligo); 2 chứng dương cho biến thể Beta (SAF_Oligo và notSAF_Oligo), 2 chứng dương cho biến thể Gamma (BrZ_Oligo và notBrZ_Oligo), 2 chứng dương cho biến thể Delta (InD_Oligo và notInD_Oligo), 2 chứng dương cho đội biến D614G (614D_Oligo và 614G_Oligo), 2 chứng dương cho đột biến E484K (484E_Oligo và 484K_Oligo). Mã số truy cập trên NCBI gene bank để từ đó thiết kế trình tự các mồi, probe và chứng [+] được trình bày trong bảng 1 trong phần phụ lục. Tất cả các mồi, probe và chứng dương đều được đặt tổng hợp từ hãng Macrogen của Hàn Quốc. Bộ xét nghiệm real-time PCR được thiết kế thành 3 multiplex real-time PCR. Multiplex A để phát hiện và định lượng SARS-CoV-2, phát hiện biến thể Alpha và phát hiện chứng nội. Multiplex B để phát hiện biến thể Beta, biến thể Gamma và biến thể Delta. Multiplex C để phát hiện các đột biến D614G và E484K. Các Multiplex real-time PCR được pha từ AgPath-ID™ One-Step RT-PCR (ThermoFisher, Mỹ), gồm 2X RT-PCR buffer, 25X RT-PCR enzyme, Nuclease free water. Công thức pha được trình bày trong bảng 2 ở phần phụ lục.
2. Khảo sát độ nhạy của bộ xét nghiệm khi thử trên các mẫu chứng dương
Các mẫu chứng [+] ở dạng đông khô được pha trong TE 1X để có hàm lượng 100pm. Sau đó pha loãng tiếp cũng trong TE 1X theo hệ số pha loãng 10 để có các độ pha loãng từ 10-9 đến 10-15 tương ứng với hàm lượng 10-1 fm/µl đến 10-7 fm/µl. Chứng [-] được sử dụng là dung dịch TE 1X. Cho 5µl các độ pha loãng của chứng [+] và chứng [-] vào các MPL A, B và C tương ứng. Thể tích của các MPL A, B và C là 15µl, như vậy tổng thể tich của mỗi phản ứng là 20µl. Bảng 3, 4 và 5 (phần phụ lục) trình bày cách cho các chứng [+] và chứng [-] vào các MPL A, B và C.
Sau khi cho các chứng [+] và chứng [-] vào các MPL A, B và C; ly tâm lắng rồi cho vào máy real-time PCR của Biorad (CFX96 touch screen, Mỹ). Chọn 4 kênh màu FAM, HEX, TexasRED và CY5. Chạy chương trình one-step RT real-time PCR gồm các bước sau: (i) Đầu tiên là chương trình phiên mã ngược ở 45oC trong 10 phút; (ii) Sau đó bất hoạt enzyme phiên mã ngược và kích hoạt enzyme hot-start Taq polymerase ở 95oC trong 10 phút; (iii) Cuối cùng chạy chương trình real-time PCR trong 40 chu kỳ với 2 bước nhiệt độ 95oC trong 15 giây để biến tính và 60oC trong 01 phút để nhân bản và phát hiện trình tự đích
3. Khảo sát khả năng phát hiện các biến thể SARS-CoV-2 trên các mẫu thật
Các mẫu thật gồm các mẫu tách chiết RNA đã xác định SARS-CoV-2 dương tính và một số mẫu thật là các mẫu thử đã được làm xét nghiệm RT-PCR và xác định là [+] với SARS-CoV-2. Với các mẫu là các tách chiết RNA thì lấy trực tiếp dịch tách chiết này để thực hiện One-step RT real-time PCR. Với các mẫu thật thì tiến hành tách chiết RNA bằng bộ thuốc thử NKDNARNAprep MAGBEAD của công ty Nam Khoa thực hiện trên máy tách chiết tự động. Các tách chiết RNA được cho vào MPL-A, MPL-B và MPL-C với thể tích 5µl cho mỗi mix chứa 15µl One-step RT real-time PCR mix. Ly tâm lắng các MPL rồi cho vào buồng ủ nhiệt của máy real-time PCR và chạy chương trình one-step RT real-time PCR như trình bày ở phần trên. Để khảo sát độ nhạy phát hiện các biến thể SARS-CoV-2 trên mẫu thật, tiến hành pha loãng các tách chiết RNA từ các mẫu thật được xác định [+] với SARS-CoV-2 rồi cho vào các MPL-A, MPL-B và MPL-C, chạy chương trình one-step RT real-time PCR rồi ghi nhận kết quả để tìm độ pha loãng cao nhất mà vẫn còn có thể phát hiện gen E , các biến thể, hai đột biến D614D và E484K. Với các mẫu đã có kết quả xác định được các biến thể của SARS-CoV-2, chọn những mẫu có kết quả real-time phát hiện sớm tức là có nồng độ vi rút cao rồi thực hiện giải trình tự toàn bộ gen S. So sánh kết quả giải trình tự với kết quả real-time trong xác định các biến thể SARS-CoV-2.
Kết quả nghiên cứu
1. Độ nhạy của bộ xét nghiệm khi thử trên các mẫu chứng dương
Độ nhạy trong phát hiện gen E: Bảng 6 (phần phụ lục) trình bày kết quả độ nhạy trong phát hiện gen E. Kết quả cho thấy độ nhạy trong phát hiện SARS-CoV-2 là 10-6 fm/µl (1-10 copies cho một thể tích dịch tách chiết RNA cho vào ống phản ứng)
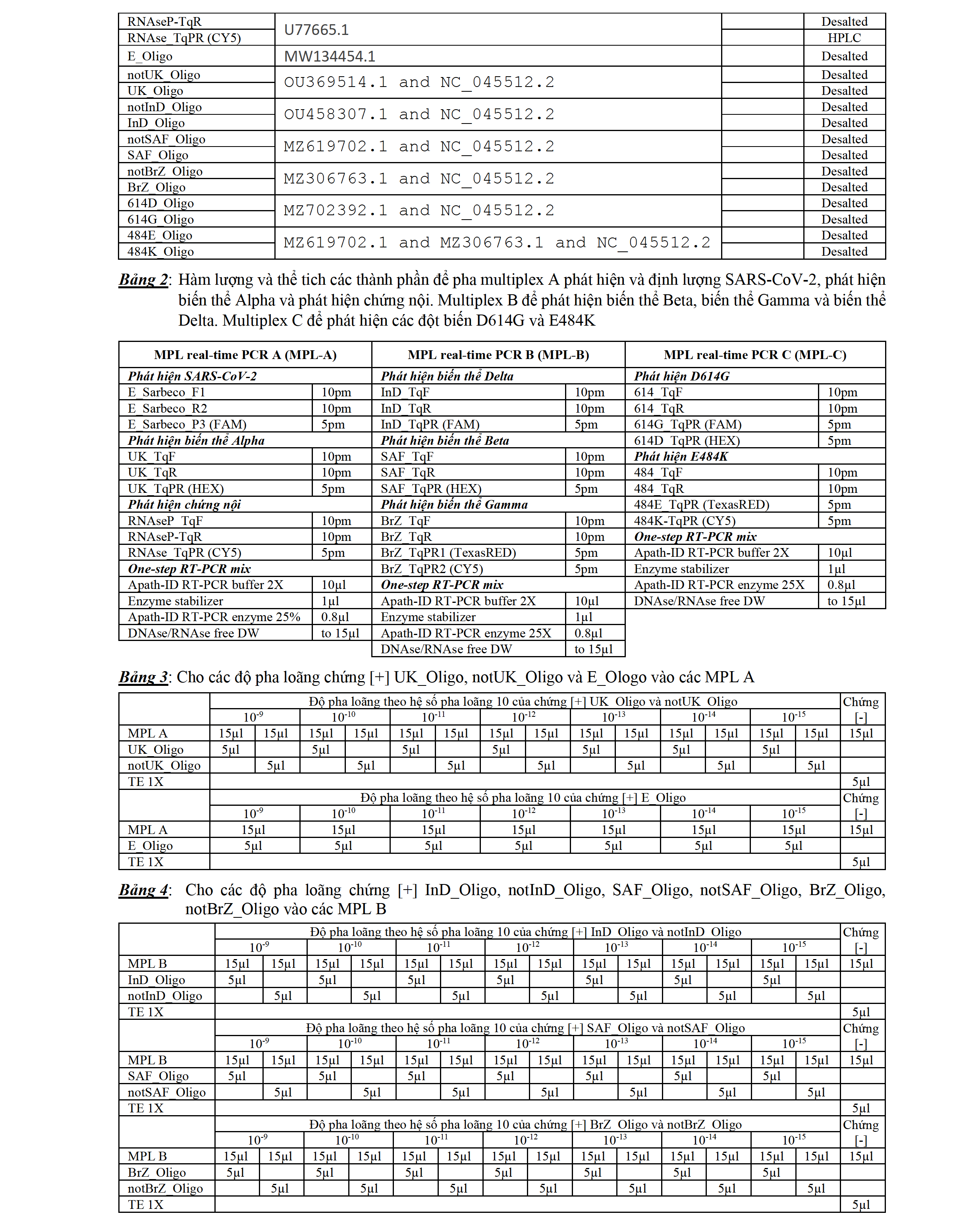
Độ nhạy trong phát hiện biến thể Alpha (UK), Delta (InD), Beta (SAF) và Gamma (BrZ): Bảng 7 (phần phụ lục) trình bày kết quả độ nhạy trong phát hiện biến thể Alpha. Kết quả cho thấy cho đến ở hàm lượng chứng [+] là 10-6 fm/µl (1-10 copy cho một thể tích dịch tách chiết RNA cho vào ống phản ứng) vẫn có thể phân biệt được biến thể UK (không cho tín hiệu khuếch đại ở kênh HEX) với không phải biến thể UK (cho tín hiệu khuếch đại ở kênh HEX). Bảng 8 (phần phụ lục) trình bày kết quả độ nhạy trong phát hiện biến thể Delta. Kết quả cho thấy cho đến ở hàm lượng chứng [+] là 10-5 fm/µl (10-100 copies cho một thể tích dịch tách chiết RNA cho vào ống phản ứng) vẫn có thể phân biệt được biến thể InD (không cho tín hiệu khuếch đại ở kênh FAM) với không phải biến thể InD (cho tín hiệu khuếch đại ở kênh FAM). Bảng 9 (phần phụ lục) trình bày kết quả độ nhạy trong phát hiện biến thể Beta. Kết quả cho thấy cho đến ở hàm lượng chứng [+] là 10-6 fm/µl (1-10 copy cho một thể tích dịch tách chiết RNA cho vào ống phản ứng) vẫn có thể phân biệt được biến thể SAF (không cho tín hiệu khuếch đại ở kênh HEX) với không phải biến thể SAF (cho tín hiệu khuếch đại ở kênh HEX). Bảng 10 (phần phụ lục) trình bày kết quả độ nhạy trong phát hiện biến thể Gamma. Kết quả cho thấy cho đến ở hàm lượng chứng [+] là 10-6 fm/µl (1 copy cho một thể tích dịch tách chiết RNA cho vào ống phản ứng), vẫn có thể phân biệt được biến thể BrZ (Ct của CY5 sớm hơn TexasRED) với không phải biến thể BrZ (Ct của CY5 không sớm hơn TexasRED)
Độ nhạy trong phát hiện các đột biến D614G và E484K: Bảng 11 (phần phụ lục) trình bày kết quả độ nhạy trong phát hiện đột biến D614G. Kết quả cho thấy cho đến ở hàm lượng chứng [+] là 10-5 fm/µl (10-100 copy cho một thể tích dịch tách chiết RNA cho vào ống phản ứng), vẫn có thể phân biệt được có đột biến D614G (Ct của FAM sớm hơn HEX) với không có đột biến D614G (Ct của FAM không sớm hơn HEX). Bảng 12 (phần phụ lục) trình bày kết quả độ nhạy trong phát hiện đột biến E484K. Kết quả cho thấy cho đến ở hàm lượng chứng [+] là 10-5 fm/µl (10-100 copy cho một thể tích dịch tách chiết RNA cho vào ống phản ứng), vẫn có thể phân biệt được có đột biến E484K (Ct của CY5 sớm hơn TexasRED) với không có đột biến E484K (Ct của CY5 không sớm hơn TexasRED)
2. Kết quả thử nghiệm trên các mẫu thật
Đọc kết quả trên MPL A để (a1) Phát hiện và định lượng gen E dựa vào tín hiệu khuếch đại của kênh FAM; (a2) Phát hiện biến thể Alpha dựa vào sự thất bại của tín hiệu khuếch đại kênh HEX; (a3) Phát hiện chứng nội đựa vào tín hiệu khuếch đại kênh CY5. Đọc kết quả trên MPL B để: (b1) Phát hiện biến thể Delta dựa vào sự thất bại của tín hiệu khuếch đại kênh FAM; (b2) Phát hiện biến thể Beta dựa vào sự thất bại của tín hiệu khuếch đại kênh HEX; (b3) Phát hiện biến thể Gamma dựa vào sự xuất hiện của tín hiệu khuếch đại kênh CY5 sớm hơn so với tín hiệu khuếch đại kênh TexasRED. Bảng 13 trình bày cách đọc kết quả để phát hiện các biến thể trên MPL A và MPL B, các kết quả ngoài kết quả trình bày trong bảng 13 không cho phép xác định biến thể. Đọc kết quả trên MPL C để: (c1) Phát hiện có đột biến D614G dựa vào sự xuất hiện của tín hiệu khuếch đại kênh FAM sớm hơn tín hiệu khuếch đại kênh HEX; (c2) Phát hiện có đột biến E484K dựa vào sự xuất hiện của tín hiệu kênh CY5 sớm hơn tín hiệu khuếch đại kênh TexasRED.
Kết quả trên các mẫu lưu lấy từ TP. HCM: Có 8 mẫu lưu RNA lấy vào tháng 4 năm 2020, kết quả có 5 mẫu âm nghiệm có lẽ do hàm lượng RNA thấp và đã bị hủy trong quá trình lưu trữ, 3 mẫu cho kết quả không phải các biến thể Alpha, Beta, Gamma và Delta, không có đột biến D614G và đột biến E484K. Đặc điểm của các mẫu này là có 1 mẫu cho chu kỳ ngưỡng sớm là 22.93, 2 mẫu cón lại cho chu kỳ ngưỡng trễ là 30.90 và 33.54. Có 23 mẫu lưu RNA lấy vào tháng 6 năm 2020, kết quả tất cả đều là biến thể Delta, có đột biến D614G và không có đột biến E484K. Đặc điểm của các mẫu này là có chu kỳ ngưỡng gen E sớm, sớm nhất là 14.2 còn trễ nhất là 31.4 với đa số là dưới 25. Kết quả được trình bày trong bảng 14 (phần phụ lục).
Kết quả trên các mẫu lưu lấy từ Quảng Nam: Có 8 mẫu lưu RNA lấy vào tháng 7 năm 2020, kết quả có 5 mẫu âm nghiệm có lẽ do hàm lượng RNA thấp và đã bị hủy trong quá trình lưu trữ, 3 mẫu cho kết quả không phải các biến thể Alpha, Beta, Gamma và Delta, có đột biến D614G nhưng không có đột biến E484K. Đặc điểm các mẫu mày là có 1 mẫu Ct 31.84, 1 mẫu Ct 25.54 và 1 mẫu Ct 27. 68. Có 12 mẫu lưu RNA lấy vào tháng 6 năm 2021, kết quả cho thấy có 7 mẫu âm nghiệm do hàm lượng RNA thấp, 4 mẫu là biến thể Alpha có đột biến D614G và không đột biến E484K, 1 mẫu không phải các biến thể Alpha, Beta, Gamma và Delta, có đột biến D614G và nhưng không có đột biến E484K. Kết quả được trình bày trong bảng 15 (phần phụ lục).
Qua các kết quả thử nghiệm trên các mẫu thật dù số lượng không nhiều nhưng cũng có thể có một số nhận định sau: (i) Ngay từ đầu dịch thì TP. HCM chỉ có chủng nguyên thủy, chưa đột biến D614G cũng như E484K, nhưng hiện nay tại TP. HCM đang lưu hành biến thể Delta có đột biến D614G nhưng vẫn chưa có đột biến E484K và tải lượng virus phát hiện được là khá cao. (ii) Tại Quảng Nam thì vụ dịch vào tháng 7 năm 2020 là do chủng đột biến D614G, nhưng hiện nay với các mẫu của tháng 6 năm 2021 thì các chủng phát hiện được là biến thể Alpha có đột biến D614G và chưa có đột biến E484K và đặc điểm chung là tải lượng virus không cao lắm. Các biểu đồ 1 đến 4 (phần phụ lục) minh họa các kết quả phát hiện các biến thể và các đột biến
So sánh với kết quả giải trình tư: Dựa trên kết quả MPL real-time PCR, chọn một mẫu biến thể Alpha (PCT-03), một mẫu biến thể Delta (P7), một mẫu không đột biến (P29) và 1 mẫu chỉ có đột biến D614G (C11) là những mẫu có Ct của gen E sớm nhất để thực hiện giải trình tự trực tiếp. Kết quả các đột biến phát hiện được qua giải trình tự được trình bày trong hình 1 và bảng 16.
Độ nhạy của xét nghiệm phát hiện các biến thể và các đột biến D614G và E484K: Phân tích chu kỳ ngưỡng của các đường biểu diễn khuếch đại trên các MPL-A, MPL-B, MPL C được cho vào các độ pha loãng tách chiết RNA của các mẫu P7 (biến thể Delta) và PCT-03 (biến thể Alpha) để xác định độ nhạy trong phát hiện gen E, các biến thể Alpha và Delta, đột biên D614G và E484K. Độ nhạy trong phát hiện gen E là độ pha loãng cao nhất hay số copy thấp nhất của vi rút mà vẫn còn tín hiệu khuếch đại kênh FAM trên MPL-A. Độ nhạy trong phát hiện các biến thể là độ pha loãng cao nhất hay số copy thấp nhất của vi rút mà vẫn còn có thể phân biệt được biến thể khi đọc kết quả trên MPL-A và MPL-C. Ví dụ cụ thể trong bảng 17 (phần phụ lục) độ nhạy phát hiện được biến thể Delta là 10 copy vì ở hàm lượng vi-rút này vẫn biết được mẫu không phải là biến thể Alpha (có tín hiệu khuếch đại trên kênh HEX ở MPL-A), không phải là biến thể Beta (có tín hiệu khuếch đại kênh HEX ở MPL-B) và cũng không phải là biến thể Gamma (Ct của TexasRED sớm hơn CY5 ở MPL-B), còn ở hàm lượng thấp hơn thì dù vẫn biết được không phải là biến thể Alpha, không phải là biến thể Beta nhưng không thể phân biệt có phải là biến thể Delta hay biến thể Gamma vì kênh FAM không có tín hiệu khuếch đại và kênh TexasRED/CY5 cũng không có tín hiệu khuếch đại. Độ nhạy của phát hiện đột biến D614G là độ pha loãng cao nhất hay số copy thấp nhất của vi rút mà trên MPL-C vẫn phân biệt được là có đột biến D614G (Ct kênh FAM sớm hơn kênh HEX) hay không có đột biến D614G (Ct kênh HEX sớm hơn kênh FAM). Độ nhạy của phát hiện đột biến E8814K là độ pha loãng cao nhất hay số copy thấp nhất của vi rút mà trên MPL-C vẫn phân biệt được là có đột biến E484K (Ct kênh CY5 sớm hơn kênh TexasRED) hay không có đột biến D614G (Ct kênh TexasRED sớm hơn kênh CY5). Phân tích kết quả trên bảng 17 cho thấy độ nhạy trong phát hiện Gen E là 1 copy, biến thể Delta là 10 copy, đột biến D614G là 10 copy và E484K cũng là 10 copy. Độ nhạy phát hiện gen E, phát hiện biến thể Alpha, đột biến D614G và E484K được trình bày trong bảng 18 (phần phụ lục). Phân tích kết quả cho thấy độ nhạy trong phát hiện Gen E là 1 copy, biến thể Alpha là 100 copy, đột biến D614G là 10 copy và E484K cũng là 10 copy. Ngoài ra tách chiết RNA của mẫu P29 được ghi nhận không có đột biến cũng được pha loãng thành nhiều độ pha loãng và được cho vào các MPL-A, MPL-B và MPL-C để phân tích độ nhạy phát hiện gen E cũng như D614G và E484K. Kết quả được trình bày trong bảng 19 (phần phụ lục). Phân tích kết quả trên bảng 19 cho thấy độ nhạy trong phát hiện Gen E là 1 copy, phát hiện không phải Alpha, Beta, Gamma và Delta cũng là 1 copy. Độ nhạy trong phát hiện không có đột biến D614G và E484K là dưới 1 copy.
Bàn luận
WHO đã có những khuyến nghị về các kỹ thuật nên được sử dụng để phát hiện các biến thể của SARS-CoV-2, dĩ nhiên kỹ thuật giải trình tự toàn bộ genome bằng phương pháp giải trình tự thế hệ mới (Next Generation Sequencing) hay bằng phương pháp Sanger, thậm chí chỉ cần giải trình tự toàn bộ gen S là cũng đủ để được xem là chuẩn vàng[10]. Tuy nhiên để có thể theo dõi kịp thời sự thay đổi của các biến thể trong một vùng dịch tễ tại các phòng xét nghiệm chẩn đoán hay chống dịch thì kỹ thuật real-time PCR được xem là thích hợp nhất. Hiện nay cũng đã có một số nghiên cứu[11-17] cũng như một số bộ kit thương mại[18-21] áp dụng kỹ thuật real-time PCR phát hiện các đột biến và các biến thể khác nhau của SARS-CoV-2, tuy nhiên vẫn còn rất hiếm các nghiên cứu hay các kit thương mại dành để phát hiện biến thể Delta và cũng chưa có một nghiên cứu phát hiện một lúc 4 biến thể lây lan nhanh như công trình nghiên cứu này.
Các công trình hay các kit thương mại hiện nay thường nhắm mục tiêu là phát hiện các đột biến rồi dựa vào sự phát hiện các đột biến để phân biệt được các biến thể. Các kỹ thuật real-time được sử dụng rất đa dạng, có thể là phân tích đường cong chảy của các sản phẩm khuếch đại, có thể là Taqman real-time PCR phát hiện các phát hiện các đa hình đơn nucleotide (SNP), có thể là real-time PCR dựa trên kẹp phân tử (molecular-clamping based real-time PCR) là kỹ thuật sử dụng thêm trình tự có xeno nucleic acid (XNA) để khóa trình tự không đột biến[21]…
Trong nghiên cứu này, chúng tôi không cần phải phát hiện nhiều đột biến mà chỉ cần phát hiện các đột biến đặc trưng cho từng biến thể để dựa vào đó mà phát hiện các biến thể. Chính nhờ vậy mà chỉ cần có hai MPL real-time PCR A và B là đủ để phát hiện 4 biến thể lây lan nhanh nhất hiện nay, đó là biến thể Alpha, Beta, Gamma và Delta. Ngoài ra, có thêm MPL real-time PCR C để phát hiện hai đột biến D614G và E484K cũng có vai trò rất quan trọng. Đột biến D614G là đột biến xuất hiện ban đầu và hiện diện trong tất cả các biến thể của SARS-CoV-2 hiện nay, nếu không có đột biến này thì chắc chắn vi rút sẽ không có khả năng lây lan nhanh. Phát hiện đột biến này giống như một chứng nội cho sự phát hiện các biến thể vì nếu phát hiện được biến thể mà không có đột biến D614G thì có thể có một sai lầm hay một bất thường nào đó. Đột biến E484K là một trong các đột biến có vai trò giúp cho vi rút kháng được khả năng trung hòa của các kháng thể đặc hiệu dùng trong điều trị người bị COVID-19 nặng và cũng có thể có nguy cơ giúp vi rút kháng được vắc xin. Đó chính là hai lý do để bộ kit phát hiện các biến thể Alpha, Beta, Gamma và Delta phải có thêm MPL C.
Tình hình dịch COVID-19 hiện nay trên thế giới là rất phức tạp, dịch bùng phát tại các quốc gia Nam Á và Đông Nam Á mà tác nhân là biến thể Delta xuất phát từ Ấn Độ. Tại các nước mà độ bao phủ vắc xin đã đủ rộng để tạo được miễn dịch cộng đồng thì dịch COVID-19 vẫn tái bùng phát vì biến thể Delta, mặc dù ở các quốc gia này số người bị COVID-19 nặng hay bị tử vong vì COVID-19 không còn cao như là trong giai đoạn không có vắc xin. Tại Việt Nam, cũng do biến thể Delta mà dịch COVID-19 hiện nay đang bùng phát mạnh tại TP. Hố Chí Minh, rồi các tỉnh thành Nam Bộ; Nguy cơ biến thể Delta lan ra các tỉnh thành miền Trung, Hà Nội và các tỉnh khác là khó tránh khỏi. Dĩ nhiên giải pháp để thoát khỏi dịch COVID-19 là phải biến COVID-19 thành một bệnh cảm cúm thông thường bằng cách chích vắc xin càng nhiểu người càng tốt để đạt được miễn dịch cộng đống. Tuy nhiên với SARS-CoV-2, một tác nhân virus mà các đột biến giúp hình thành các biến thể lây lan nhanh hơn và gây bùng phát dịch không chỉ là thực tế mà còn là khả năng. Chính vì vậy việc nhanh chóng phát hiện các biến thể cũng như các bất thường trong phát hiện các biến thể là rất cần thiết để giúp truy vết nhanh nguy cơ xuất hiện các ổ dịch cũng như bùng phát dịch mới. Dĩ nhiên tại các trung tâm nghiên cứu quốc tế về SARS-CoV-2 thì việc giải trình tự các mẫu lấy từ bệnh nhân được phát hiện [+] với SARS-CoV-2 là một nghiên cứu được tiến hành liên tục. Tuy nhiên với các phòng thí nghiệm chẩn đoán hay chống dịch thì với phương tiện real-time vẫn có thể phát hiện được sự lưu hành của các biến thể hiện hành và cũng có thể báo trước được sự thay đổi biến thể nếu có trang bị thêm các thuốc thử phát hiện các biến thể. Bộ thuốc thử được phát triển trong công trình nghiên cứu này chính là để đáp ứng cho nhu cầu thực tế đó.
Kết luận
Công trình nghiên cứu này có thể được xem là một trong rất ít nghiên cứu sử dụng real-time PCR phát hiện được 4 biến thể lây lan nhanh nhất hiện nay. Do khả năng tiếp cận được các nguồn mẫu lưu còn hạn chế nên chúng tôi chỉ thử nghiệm trên một số ít mẫu lưu thôi. Tuy nhiên với các kết quả ghi nhận được chúng tôi cũng có thể có một nhận định về diễn tiến lưu hành các biến thể tại Việt Nam từ khi khởi phát dịch vào tháng 4 năm 2020 đến nay. Khi mới bắt đầu dịch COVID-19 thì Việt Nam lưu hành dòng SARS-CoV-2 nguyên thủy và có cũng có một số ít có đột biến D614G. Tuy nhiên đến tháng 7 năm 2020 thì chủng SARS-CoV-2 có đột biến D614G đã bắt đầu thay thế chủng nguyên thủy. Các đợt bùng phát dịch này vẫn còn lẻ tẻ vì hầu hết là từ các khách Âu Mỹ đến Việt Nam chưa lây lan ra cộng đồng. Tuy nhiên đến cuối năm 2020, dịch bùng phát tại Bắc Giang và chủng lưu hành theo các thông tin báo chí là biến thể Alpha. Từ lúc này Việt Nam đã bắt đầu khá vất vả để đối phó biến thể này. Hiện nay biến thể đang lưu hành tại TP. HCM là biến thể Delta và như chúng ta đã thấy việc đối phó với dịch COVID-19 gây ra do biến thể Delta gây ra là cực kỳ khó khăn; nguy cơ biến thể sau khi lây lan về các tỉnh thành Nam Bộ sẽ tiếp tục lây lan đến các tỉnh thành Trung và Bắc là rất có khả năng xãy ra. Nếu có điều kiện, chúng tôi sẽ thử nghiệm trên các mẫu lưu tại các tỉnh thành và như vậy là sẽ có một bản đồ lịch sử dịch tễ chi tiết hơn về sự lưu hành của các biến thể SARS-CoV-2 tại Việt Nam.
Tài liệu tham khảo
1. Ewen Callaway. (2020). Making Sense of Coronavirus Mutations. Nature. 585:174-177
2. Thuong V. Nguyen et al. (2021). Genome‐wide analysis of SARS‐CoV‐2 strains circulating in Vietnam: Understanding the nature of the epidemic and role of the D614G mutation. J Med Virol. 2021;1–6.
3. Tam Thi Nguyen et al. (2020). Genetic diversity of SARS-CoV-2 and clinical, epidemiological characteristics of COVID-19 patients in Hanoi, Vietnam. PLOS ONE | https://doi.org/10.1371/journal.pone.0242537 November 17, 2020
3. CDC. (2021). SARS-CoV-2 Variant Classifications and Definitions. Updated June 15, 2021.
4. Rambaut A, Loman N, Pybus O, Barclay W, Barrett J, Carabelli A,et al. (18 December 2020). "Preliminary genomic characterisation of n emergent SARS-CoV-2 lineage in the UK defined by a novel set of spike mutations" https://virological.org/t/preliminary-genomic-characterisation-of-an-emergent-sars-cov-2-lineage-in-the-uk-defined-by-a-novel-set-of-spike-mutations/563
5. Horby, Peter; Barclay, Wendy; Huntley, Catherine (13 January 2021). NERVTAG paper: brief note on SARS-CoV-2 variants (https://www.gov.uk/government/publications/nervtag-brief-note-on-sars-cov-2-variant s-13-january-2021).
6. "Confirmed cases of COVID-19 variants identified in UK" (https://www.gov.uk/government/news/confirmed-cases-of-covid-19-variants-identified-in-uk)
7. Horby, Peter; Barclay, Wendy; Gupta, Ravi; Huntley, Catherine (27 January 2021). NERVTAG paper: note on variant P.1 (https://www.gov.uk/government/publications/nervtag-note-on-variant-p1-27-january-2021)
8. Azad, Arman (17 March 2021). "Coronavirus strains first detected in California are officially 'variants of concern,' CDC says" (https://edition.cnn.com/2021/03/17/health/california-variants-of-concern-cdc/index.html).
9. SARS-CoV-2 variants of concern and variants under investigation in England, technical briefing10 (https://assets.publishing.service.gov.uk/government/uploads/system/uploads/attachment_data/file/ 984274/Variants_of_Concern_VOC_Technical_Briefing_10_England.pdf)
10. ECDC and WHO. (2021). Methods for the detection and identification of SARS-CoV-2 variants. March 2021
11.https://www.thermofisher.com/vn/en/home/clinical/clinical-genomics/pathogen-detection-solutions/real-time-pcr-research-solutions-sars-cov-2/mutation-panel.html
12. E. ´Alvarez-Argüelles et Al. (2021). A novel single nucleotide polymorphism assay for the detection of N501Y SARS-CoV-2 variants. Journal of Virological Methods 294 (2021) 114143
13. Octavio Patricio Garcı´a-Gonza´ lez; José Francisco Muñoz-Valle et al. (2021). RT-qPCR Assays for Rapid Detection of the N501Y, 69-70del, K417N, and E484K SARS-CoV-2 Mutations: A Screening Strategy to Identify Variants With Clinical Impact. Frontiers in Cellular and Infection Microbiology. Volume 11 | Article 672562
14. Ariel Kushmaro et al. (2021). Direct RT-qPCR assay for SARS-CoV-2 variants of concern (Alpha, B.1.1.7 and Beta, B.1.351) detection and quantification in wastewater. Environmental Research 201 (2021) 111653
15. R. Fauver, Nathan D. Grubaugh et al. (2021). Multiplex qPCR discriminates variants of concern to enhance global surveillance of SARS-CoV-2. PLOS Biology May 7, 2021
16. G La Rosa et al. (2021). Rapid screening for SARS-CoV-2 variants of concern in clinical and environmental samples using nested RT-PCR assays targeting key mutations of the spike protein. Water Research 197 (2021) 117104.
17. Maidar Jamba, Michael Y. Sha et al. (2021). A Rapid SARS-CoV-2 Variant Detection 1 by Molecular-Clamping Based RT-qPCR. medRxiv preprint doi: https://doi.org/10.1101/2021.04.01.21254484; this version posted April 5, 2021.
18.https://en.vircell.com/products/sars-cov-2-variants-realtime-pcr-kit/ (Alpha, Beta, Gamma)
19. https://www.certest.es/news/new-viasure-sars-cov-2-variant-i-variant-ii-rt-pcr-kits/
20. https://www.bio-rad.com/featured/en/sars-cov-2-variants-pcr-assays.html
21. https://pentabase.com/mutations/
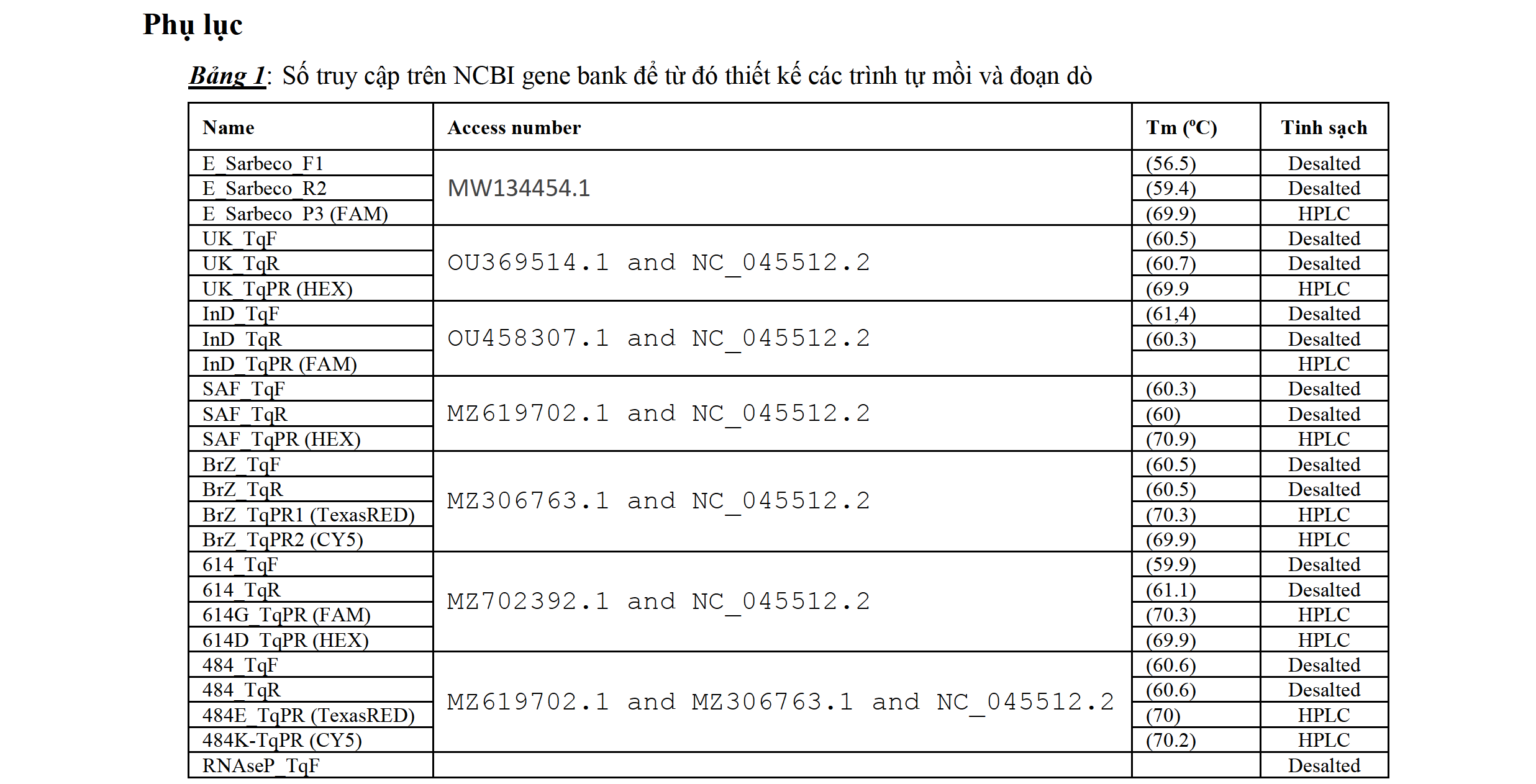




-

Chuyên gia dinh dưỡng gợi ý 7 món ăn tối tốt nhất giúp giảm mỡ và tăng cơ
Bạn muốn giảm cân nhưng không biết nên ăn gì vào buổi tối? Hãy tham khảo những thực phẩm lý tưởng giúp giảm mỡ, tăng cơ, hỗ trợ quá trình giảm cân hiệu quả.
-

9 cách đã được chứng minh để ngăn ngừa phồng rộp chân khi chạy
Phồng rộp chân có thể hình thành do ma sát quá mức khi bạn chạy. Giày không vừa chân, đổ mồ hôi quá nhiều, tập luyện quá sức, hoặc thậm chí cả tư thế chạy của bạn đều có thể góp phần gây phồng rộp. Tuy nhiên, có những biện pháp bạn có thể thực hiện để ngăn ngừa tình trạng này.
-

Chăm sóc làn da quá mức có thể gây hậu quả gì?
Việc chăm sóc làn da quá mức đôi khi mang lại tác dụng ngược, khiến làn da trở nên mệt mỏi và tổn thương. Bài viết này giúp bạn nhận diện sớm các dấu hiệu da bị quá tải và lộ trình phục hồi an toàn.
-

Luteolin - chất chống oxy hóa tiềm năng và vai trò trong một số bệnh
Luteolin là một flavonoid quý giá trong rau xanh và các thảo mộc truyền thống, nổi bật với khả năng chống viêm mạnh mẽ và bảo vệ thần kinh nhờ khả năng vượt qua hàng rào máu - não.
-

Những điều cần biết về tiếng thở khò khè
Khò khè là âm thanh phát tra từ khí phế quản do hơi thở của bạn đi qua đường thở bị co hẹp do một bệnh nào đó của phổi. Hen suyễn và bệnh phổi tắc nghẽn mãn tính (COPD) là những nguyên nhân phổ biến nhất gay ra tiếng khò khè.
-

Giữ cân sau giảm béo: Khó hay dễ?
Giảm cân thành công là một cột mốc đáng tự hào, tuy nhiên hành trình để sở hữu thân hình khỏe mạnh chỉ mới bắt đầu. Thực tế cho thấy một tỷ lệ lớn người giảm cân đều gặp phải tình trạng tăng cân trở lại, đôi khi còn trầm trọng hơn mức ban đầu.
-

Yoga và thiền định: Giải tỏa căng thẳng, tâm thần an tĩnh
Thiền định được công nhận có tác dụng chăm sóc sức khỏe tinh thần và tìm lại sự cân bằng trong cuộc sống. Trong nhịp sống hối hả, căng thẳng là điều khó tránh khỏi. Tuy nhiên, yoga và thiền định là hai phương pháp hiệu quả để giảm căng thẳng và nuôi dưỡng sự an tĩnh trong tâm hồn. Bài viết này sẽ khám phá cách yoga và thiền định mùa thu giúp cải thiện sức khỏe tinh thần, đồng thời hướng dẫn bạn cách kết hợp chúng vào cuộc sống hàng ngày để đạt được trạng thái cân bằng.
-

Uống trà xanh với chanh ngon, tốt nhưng 3 nhóm người nên thận trọng
Trà xanh pha với chanh là một thức uống ngon và sảng khoái. Tuy nhiên sự kết hợp này có thực sự có lợi cho sức khỏe tất cả mọi người không?